データ解析のための統計モデリング入門 GLMのモデル選択 読書メモ2
2017年 02月 28日
このブログ記事は『データ解析のための統計モデリング入門』(久保拓弥 著、岩波書店)という、とても分かりやすい統計モデリングの入門書を、さらに分かりやすくするために読書メモをまとめたものです。
今回は第4章、GLMのモデル選択についてのまとめの二回目です。
この章ではバイアスやAICについて説明がされています。
データを説明するモデルとして様々なモデルが考えられる時に、データへのあてはまりの良さだけを基準にモデルを選ぶことはできません。
それを理解するために、ポアソン回帰で推定されるパラメータがどれくらい真の値からずれるのかをヒストグラムにしてくれるコードを用意しました。
コードはRで書きました。
createHistogram <- function(n) {
lambdas <- vector(length = 1000)
for (i in 1:1000) {
y <- rpois(n, lambda = 4)
data <- data.frame(y)
fit <- glm(formula = y ~ 1, family = poisson, data = data)
lambdas[i] <- exp(fit$coefficients[1])
}
title <- paste("Number of data =", n)
hist(lambdas[lambdas <= 10], breaks = seq(0, 10, by = 0.01), main = title, xlab = "Estimeted lambda")
}
for (i in c(1, 5, 10, 100, 200)) {
createHistogram(i)
}実行すると平均 4 のポアソン分布から n 個のサンプルを取って、ポアソン回帰をするということを1000回繰り返して、推定結果のヒストグラムを描きます。
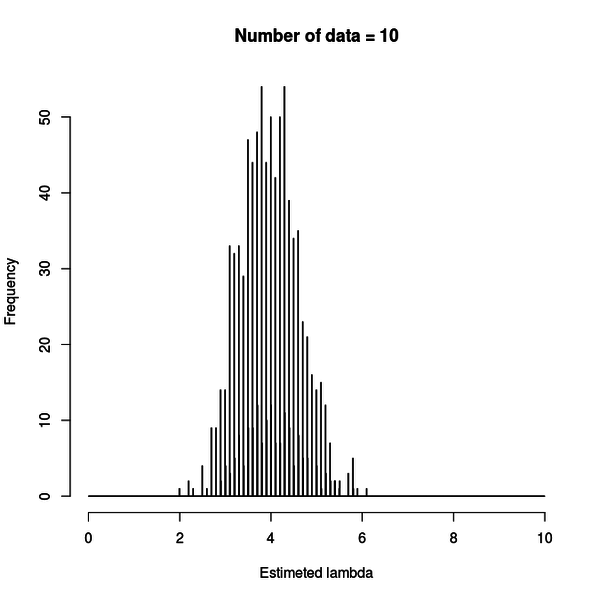
リンク関数は定数です。n が 10 の時は下図のようになります。
n が 100 の時は下図のようになります。
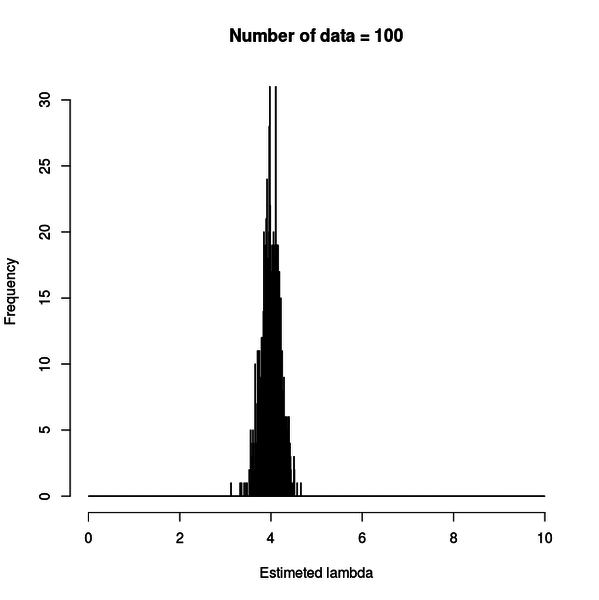
推定結果が真の値である 4 の周囲に分布しています。
データの数が多くなれば分布のピークは 4 に集中していきますが、データが 100 個あっても、やはりずれる時はずれるのが分かります。



