データ解析のための統計モデリング入門 一般化線形混合モデル(GLMM) 読書メモ9
2017年 06月 13日
このブログ記事は『データ解析のための統計モデリング入門』(久保拓弥 著、岩波書店)という、とても分かりやすい統計モデリングの入門書を、さらに分かりやすくするために読書メモをまとめたものです。
今回は第7章、一般化線形混合モデル(GLMM)についてのまとめの九回目です。
この章では複数の分布を混ぜて使う、一般化線形混合モデルついて説明がされています。
その中で二項分布とと正規分布を混ぜて使う一般化線形混合モデルについて説明されています。
なので、サンプルデータを用意して、様々なパラメータにおける対数尤度の値をプロットしてくれるコードを用意しました。
コードはRで書きました。
dataSize <- 50
N <- 20
linkFunction <- function(b1, b2, x) {
function(r) {
1 / (1 + exp(-b1 - b2 * x - r))
}
}
dmixture <- function(b1, b2, s) {
function(y, x) {
l <- linkFunction(b1, b2, x)
sum <- 0
for (r in seq(-100, 100, by = 0.1)) {
sum <- sum + dbinom(y, N, l(r)) * dnorm(r, mean = 0, sd = s) * 0.1
}
sum
}
}
likelihood <- function(b1, b2, s) {
d <- dmixture(b1, b2, s)
function (y, x) {
d(y, x)
}
}
logLikelihood <- function(data, b1, b2) {
function (s) {
sl <- 0
ll <- likelihood(b1, b2, s)
for (i in 1:dataSize) {
sl <- sl + log(ll(data$y[i], data$x[i]))
}
sl
}
}
createData <- function() {
x <- runif(dataSize, 0, 5)
y <- vector(length = dataSize)
for (i in 1:dataSize) {
r <- rnorm(1, mean = 0, sd = 1)
l <- linkFunction(-2, 1, x[i])
y[i] <- rbinom(1, N, l(r))
}
data.frame(x, y)
}
data <- createData()
xl <- "s"
yl <- "Lilelihood"
ss <- seq(0.2, 2, by = 0.2)
b2s <- seq(-2, 2, by = 1)
legends <- paste0("b2 = ", b2s)
for (b1 in seq(-2, 2, by = 1)) {
title <- paste0("logit(q) = ", b1, " + b2 * x + r, r ~ N(0, s)")
plot(0, 0, type = "n", xlim = c(0, 2), ylim = c(-1000, 0), main = title, xlab = xl, ylab = yl)
for (i in 1:5) {
ls <- logLikelihood(data, b1, b2s[i])(ss)
lines(ss, ls, type = "l")
points(ss, ls, pch = i)
}
legend("topleft", legend = legends, pch = 1:5)
}リンク関数は logit(q) = b1 + b2 * x + r です。r は平均 0 、標準偏差 s の正規分布から生成されます。
サンプルデータを生成するには、まず 0 から 5 までの一様分布から x を、標準偏差 s = 1 の正規分布から r を生成します。
その後、b1 = -2、b2 = 1 のリンク関数を使って事象の発生確率 q を求め、試行回数 N = 20 の二項分布から値を生成しています。
なので、様々なパラメータについてモデルを試すと、b1 = -2、b2 = 1、s = 1 の時に最もデータへの当てはまりがよくなるはずです。
つまり尤度が最大になるはずです。
コードを実行すると5枚のグラフがプロットされます。
例えば次のようなグラフが得られます。
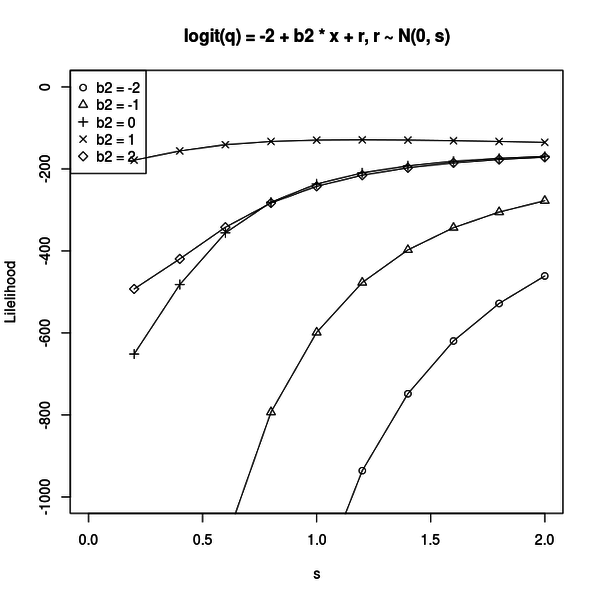
図を見ると、期待通りに b1 = -2、b2 = 1、s = 1 の場合に尤度が最大になっているのが分かります。
実際の問題では、これらのパラメータを最尤推定すればいいわけです。



