データ解析のための統計モデリング入門 マルコフ連鎖モンテカルロ(MCMC)法とベイズ統計モデル 読書メモ3
2017年 06月 27日
このブログ記事は『データ解析のための統計モデリング入門』(久保拓弥 著、岩波書店)という、とても分かりやすい統計モデリングの入門書を、さらに分かりやすくするために読書メモをまとめたものです。
今回は第8章、マルコフ連鎖モンテカルロ(MCMC)法とベイズ統計モデルについてのまとめの三回目です。
この章ではMCMCやメトロポリス法について説明がされています。
その中で、メトロポリス法によるサンプリングが解説されています。
なので、同じようにメトロポリス法でサンプリングしてみるコードを用意しました。
コードはRで書きました。
dataSize <- 20
N <- 8
data <- rbinom(dataSize, N, 0.45)
likelihood <- function (q) {
sum(log(dbinom(data, N, q)))
}
develop <- function(q) {
if (runif(1, 0, 1) < 0.5) {
q + 0.01
} else {
q - 0.01
}
}
sampling <- function(n, init) {
q <- init
ql <- likelihood(q)
samples <- vector(length = n)
i <- 1
while(i <= n) {
p <- develop(q)
pl <- likelihood(p)
r <- log(runif(1, 0, 1))
if (ql < pl || r < pl - ql) {
q <- p
ql <- pl
}
samples[i] <- p
i <- i + 1
}
samples
}
samples1 <- sampling(500, 0.3)
hist(samples1, breaks = seq(0, 1, by = 0.01), main = "500 samples", xlab = "q")
samples2 <- c(samples1, sampling(500, samples1[500]))
hist(samples2, breaks = seq(0, 1, by = 0.01), main = "1000 samples", xlab = "q")
samples3 <- c(samples2, sampling(1000, samples2[1000]))
hist(samples3, breaks = seq(0, 1, by = 0.01), main = "2000 samples", xlab = "q")
samples4 <- c(samples3, sampling(8000, samples3[2000]))
hist(samples4, breaks = seq(0, 1, by = 0.01), main = "10000 samples", xlab = "q")サンプルデータとしては、本の中でやっているのと同じように、事象の発生確率が 0.45 で、事象の試行回数は 8 の二項分布から、20 個のサンプルを取って用意しました。
なので、このデータに対して q のサンプリングをすると、だいたい 0.45 を中心とした分布が得られるはずです。
コードではサンプル数が 500、 1000、 2000、 10000 の時にヒストグラムをプロットしています。
コードを実行すると次のような図がプロットされます。
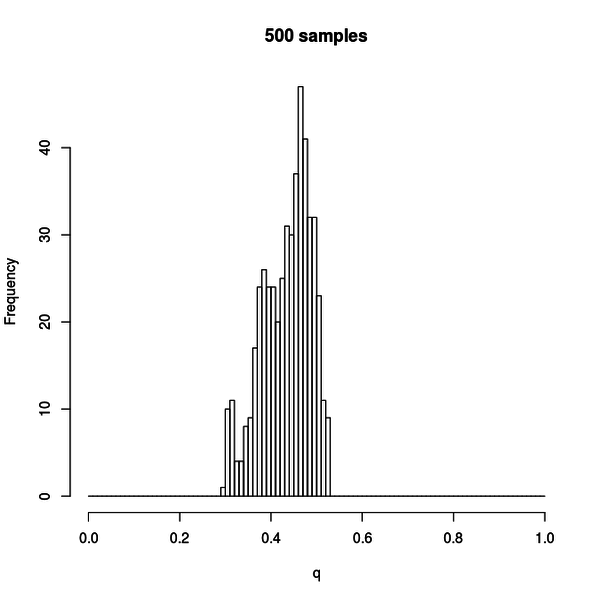
この図はサンプル数 500 の場合です。
まだヒストグラムが凸凹しているのが分かります。
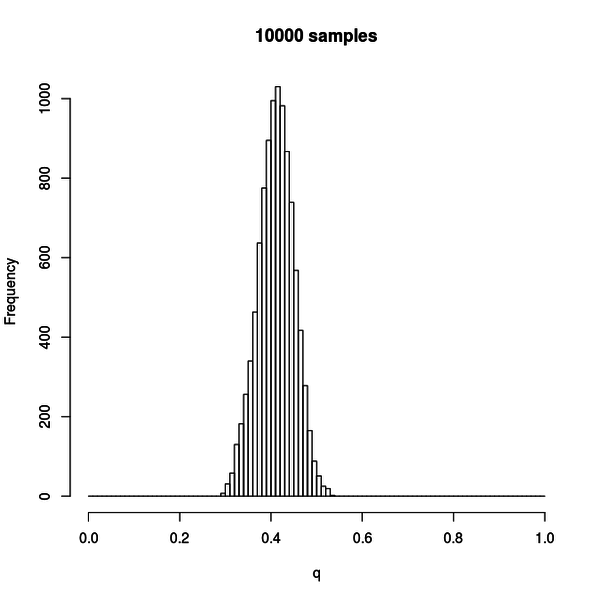
この図はサンプル数 10000 の場合です。
だいぶヒストグラムがなめらかになって、分布が収束してきているのが分かります。



